こんにちは。ほしのはやしです。
Intervention群とControl群にわけて薬剤を投与したあとの各時点での比較をしたい場合、R studioでどのように解析を行うのか、できる限りわかりやすく解説していきたいと思います!
今回扱う経時データのグラフ
まずはどのようなグラフを想定しているか共有していきます。
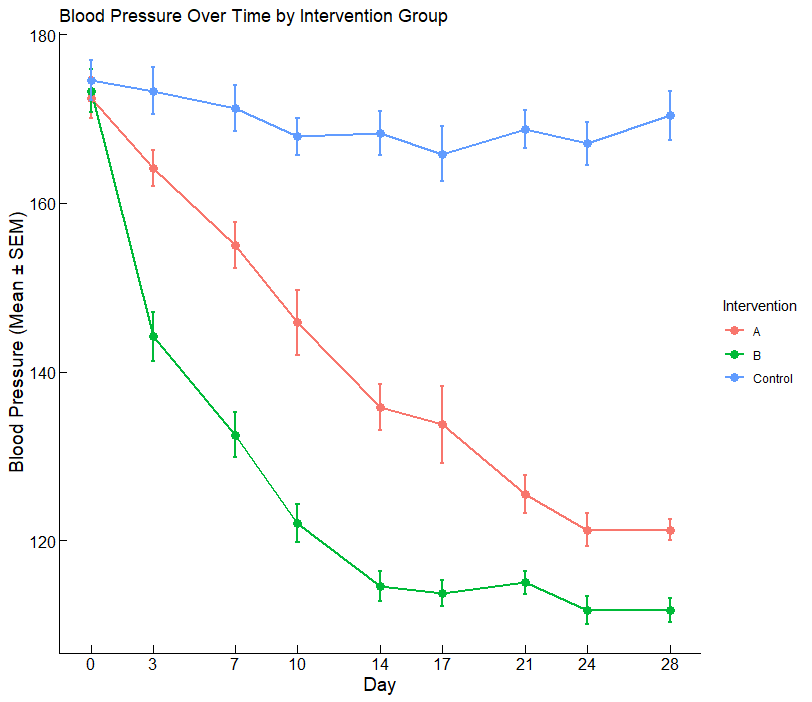
実験系でよく見るパターンのグラフです。
A, B, Control群でDay 28時点での差を見るなら、その時点をピックアップしてANOVAなりすればよいのですが、どの時点から差が出ているのか全ての時点で比較したいですよね!
本ページではその方法について具体的に解説していきます!
 星柴くん
星柴くんネットで調べてみても意外と取り扱ってるサイトないのだ
Rでの解析方法
時系列データをlong形式:長形式にする
多くの場合、以下のようにエクセルにデータを入れていると思います。
データ名:datasheet
| Subject | Intervention | Day0 | Day3 | Day7 | Day10 |
|---|---|---|---|---|---|
| 1 | A | 180 | 170 | 160 | 150 |
| 2 | B | 160 | 120 | 110 | 115 |
| 3 | Control | 165 | 155 | 160 | 170 |
| 4 | A | 170 | 160 | 150 | 140 |
| 5 | B | 175 | 155 | 135 | 120 |
| 6 | Control | 185 | 190 | 160 | 180 |
| … | … | … | … | … | … |
これを以下のように解析に適した形に変更する必要があります。
この形のことをlong形式と呼びます。
| Subject | Intervention | Day | BloodPressure |
|---|---|---|---|
| 1 | A | Day0 | 180 |
| 1 | A | Day3 | 170 |
| 1 | A | Day7 | 160 |
| 1 | A | Day10 | 150 |
| 1 | A | Day14 | 140 |
| 1 | A | Day17 | 120 |
| … | … | … | … |
このように変更するためのRスクリプトは以下になります。
library(tidyverse)
# データをlong形式に変換
long_data <- datasheet %>% pivot_longer(cols = starts_with(“Day”), names_to = “Day”, values_to = “BloodPressure”)
下記のようにDay列を数値だけをピックアップしたい場合は、以下のスクリプトを続けます。
データ名:long_data
| Subject | Intervention | Day | BloodPressure |
|---|---|---|---|
| 1 | A | 0 | 180 |
| 1 | A | 3 | 170 |
| 1 | A | 7 | 160 |
| 1 | A | 10 | 150 |
| 1 | A | 14 | 140 |
| 1 | A | 17 | 120 |
| … | … | … | … |
# 上記で作成したDay列から数値だけを取り出し、数値型に変換
long_data$Day <- as.numeric(gsub(“Day”, “”, long_data$Day))
平均値とSEMの計算をして新しいデータシートを作る
summary_data <- long_data %>%
group_by(Intervention, Day) %>%
summarise(mean_bp = mean(BloodPressure),
sem_bp = sd(BloodPressure) / sqrt(n()))
平均値とSDにしたい場合は以下のスクリプトを使用。
summary_data <- long_data %>%
group_by(Intervention, Day) %>%
summarise(mean_bp = mean(BloodPressure),
sd_bp = sd(BloodPressure))
エクセルに平均値のデータを出力したい場合は、以下になります。
library(openxlsx)
write.xlsx(summary_data, file = “test.xlsx”)
折れ線時系列グラフを作成する
パッケージggplot2を用いて、グラフを描きます。
library(ggplot2)
ggplot(summary_data, aes(x = Day, y = mean_bp, group = Intervention, color = Intervention)) +
geom_line(linewidth = 1) +
geom_point(size = 3) +
geom_errorbar(aes(ymin = mean_bp – sem_bp, ymax = mean_bp + sem_bp), size = 1, width = 0.2) +
labs(title = “Blood Pressure Over Time by Intervention Group”,
x = “Day”,
y = “Blood Pressure (Mean ± SEM)”) +
theme(axis.line = element_line(color = “black”, linewidth = 0.3, linetype = “solid”),
axis.text = element_text(color = “black”, size = 12),
axis.title = element_text(size = 14),
panel.background = element_blank(),
axis.ticks.length = unit(-2, “mm”),
axis.ticks = element_line(color = “black”, linewidth = 0.3)) +
scale_x_continuous(breaks = unique(long_data$Day))
統計解析
全ての群、全てのタイミングで正規性がある(あると考えられるような実験系の)場合は、下記のようにANOVAを用いて全てのタイミングで比較して多重性を補正します。
# 各時点ごとにANOVAと事後検定を実施
anova_results <- list()
posthoc_results <- list()
for (day in unique(long_data$Day)) {
subset_data <- long_data %>% filter(Day == day)
model <- aov(BloodPressure ~ Intervention, data = subset_data)
anova_results[[as.character(day)]] <- summary(model)
posthoc_results[[as.character(day)]] <- TukeyHSD(model)
}
# ANOVAの結果を表示
anova_results
# 事後検定の結果を表示
posthoc_results
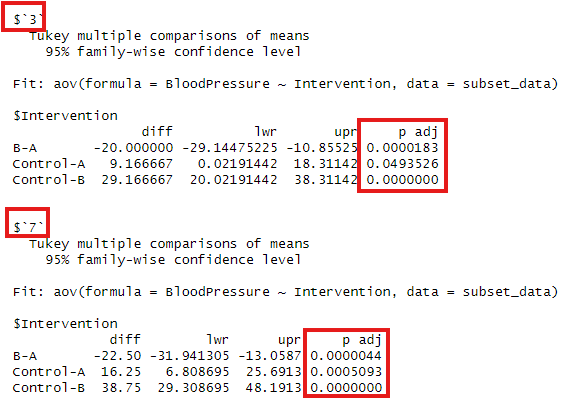

抜粋した結果ですが、左上がDay 3, Day 7で補正されたp値が右側になります。
正規性がない場合は以下の方法で計算します。今回は多重性の補正にDunn.testを用いています。
library(dunn.test)
# Day毎のKruskal-Wallis検定
kruskal_result <- long_data %>% group_by(Day) %>%
summarise(p_value = kruskal.test(BloodPressure ~ Intervention)$p.value)
# 有意な結果を持つDayを抽出
significant_days <- kruskal_result %>% filter(p_value < 0.05) %>% pull(Day)
# 各有意なDayについてDunnの手順による多重比較を実行
dunn_results <- lapply(significant_days, function(day) { dunn.test::dunn.test(long_data$BloodPressure[long_data$Day == day], long_data$Intervention[long_data$Day == day], method = “bonferroni”) })
# 結果を表示
names(dunn_results) <- paste(“Day”, significant_days)
dunn_results
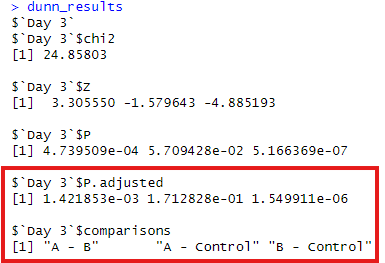

Day毎に補正されたP値が表示されます。



実験系の場合は厳密性はあまり重視されず、ANOVAが使用されていることがほとんどなのだ。。。
まとめ
経時データをグラフ化および各時点毎を統計評価する方法について解説しました!
少しでもお役に立てれば幸いです!!










コメント