こんにちは。ほしのはやしです。
棒グラフ・箱ヒゲ図にドットをつけることが最近は当たり前になってきました。
このページではドットグラフの必要性・棒グラフ/箱ヒゲ図の使い分け・R studioを使ってRで複合グラフを作る方法、の3点について解説してきます!
なぜドットグラフが必要か
棒グラフや箱ひげ図にドットグラフ(個々のデータ点をプロット)を加えることで、データの分布や特徴をより詳細に把握することができます。
ドットグラフを加えることで得られる情報
- 外れ値の確認: データセットの中に極端に大きい値や小さい値(外れ値)があるかどうかを視覚的に確認できる。
- 分布の形状: データが正規分布に従っているか、それとも歪んでいるかなどを確認できる。
- データの集まり方: データがどのあたりに集中しているのか、あるいは特定の値に偏っているのかを視覚的に確認できる。
- グループ間の比較: 複数のグループのデータを比較する際、各データ点のばらつきや重なり具合を詳細に比較できる。
何例以下ならドットを表示すべきか?
ドットグラフを表示する際のデータ数については、明確な基準はありません。
データの量や種類、分析の目的によって最適な表示方法は異なります。
一つの目安として、データ数が50個以下であればすべてのデータをプロットしても問題ないことが多いです。
棒グラフと箱ひげ図、どちらを使うべき?
数値データを視覚的に表現し、読者に情報を効果的に伝えるために、棒グラフや箱ひげ図がよく用いられます。
どのようなグラフを選ぶべきか、具体的な例を交えて解説します。
棒グラフが適しているケース
- カテゴリー別の数値の比較:
- 例:異なる治療法を受けた患者群の生存率の比較
- 説明:各治療法に対応する棒の長さで、生存率の高低を視覚的に表現できる。
- 割合の比較:
- 例:特定の疾患を持つ患者における性別比
- 説明:各性別に対応する棒の長さで、患者の性別分布を分かりやすく示せる。
- 頻度の比較:
- 例:ある薬剤の副作用の種類別の発生頻度
- 説明:各副作用に対応する棒の長さで、副作用の発生頻度の高低を比較できる。
箱ひげ図が適しているケース
- データの分布の比較:
- 例:異なる年齢層の血圧の分布
- 説明:各年齢層の血圧のばらつきや中央値を箱ひげ図で比較することで、年齢による血圧の変化を詳しく分析できる。
- 外れ値の確認:
- 例:ある検査値における異常値の有無
- 説明:箱ひげ図のひげから外れた点が外れ値として確認できる。
- 複数のグループ間の比較:
- 例:異なる治療法を受けた患者群の腫瘍サイズ
- 説明:複数の治療法における腫瘍サイズの分布を同時に比較できる。
 星柴くん
星柴くんこれを考えると、ドットプロットを合わせて表示したい場合は棒グラフよりも箱ひげ図の方が適切、と言えるね!!!



一般的にはそう言われてるやで。でも平均値を強調したいような医学論文の場合では、あえて棒グラフを使うこともよくあるんやで!
Rで箱ヒゲ図にドットプロットを重ねる方法
Rで練習用のデータ作成
まずは練習用のデータを作成します。
グラフの目的
新規薬(drug)の種類が LDL 値に与える影響を、遺伝子Aの型(group: WT と KO)ごとに比較
⇨新規薬の作用機序に遺伝子Aが関連していることを証明
データシート名:LDLdata
library(tidyverse)
# ランダムシードを設定して再現性のあるデータにする
set.seed(123)
# データフレームの作成
LDLdata <- data.frame(
group = rep(c("WT", "KO"), each = 60),
gene = rep("geneA", 120),
drug = rep(c("placebo", "A", "B", "placebo", "A", "B"), each = 20), # 順序はmutateで設定
LDL = c(
rnorm(20, mean = 120, sd = 15), # プラセボ WT
rnorm(20, mean = 80, sd = 10), # Drug A WT (有意に低い)
rnorm(20, mean = 85, sd = 10), # Drug B WT (有意に低い)
rnorm(20, mean = 115, sd = 15), # プラセボ KO
rnorm(20, mean = 112, sd = 15), # Drug A KO
rnorm(20, mean = 118, sd = 15) # Drug B KO
)
)
# グループと薬剤の順序を指定
LDLdata <- LDLdata %>%
mutate(group = factor(group, levels = c("WT", "KO")), # コントロール (WT) を左側に配置
drug = factor(drug, levels = c("placebo", "A", "B"))) # 薬剤の順序を明示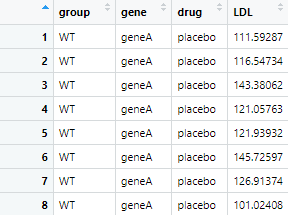

これで、「group」に遺伝子操作のグループ、「gene」に遺伝子(今回はgeneAに統一)、「drug」に薬剤、「LDL」にLDLの数値を入力したデータができました!
Rで箱ヒゲ図の作成
続いて、このデータを用いて箱ヒゲ図とドットプロットを書きます!
このときに『ggpubr』というパッケージが必要なので以下のサイトを参考にパッケージをインストールしてください。


以下にコードをお示しします。
library(ggpubr)
# ggboxplotを使用して箱ひげ図を作成
ggboxplot(
LDLdata,
x = "drug", y = "LDL", color = "group", fill = "group",
add = "jitter", # ドットプロットを追加
facet.by = "group", # "グループ"で群分け(ファセットの表示)
palette = c("white", "gray20") # 塗りつぶしの色を指定 (WT: 白, KO: 黒に近い灰色)
) +
# 枠線を黒に強制
scale_color_manual(values = c("black", "black")) +
# 有意差検定の追加
stat_compare_means(
comparisons = list(c("placebo", "A"), c("placebo", "B")),
method = "t.test",
label = "p.signif",
) +
# ラベルの設定
labs(x = "薬の種類", y = "LDL値", color = "遺伝子型", fill = "遺伝子型") +
# 見た目・レイアウトの調整
theme_classic()+
theme(
strip.background = element_blank(), # ファセットタイトルの背景を消す
strip.text = element_blank(), # ファセットタイトルのテキストを消す
axis.line = element_line(color = "black", size = 0.3, linetype = "solid"),
axis.text = element_text(color = "black", size = 12),
axis.title = element_text(size = 14),
panel.background = element_blank(), # ファセットの背景を削除
axis.ticks.length = unit(-2, "mm"),
axis.ticks = element_line(color = "black", size = 0.3),
)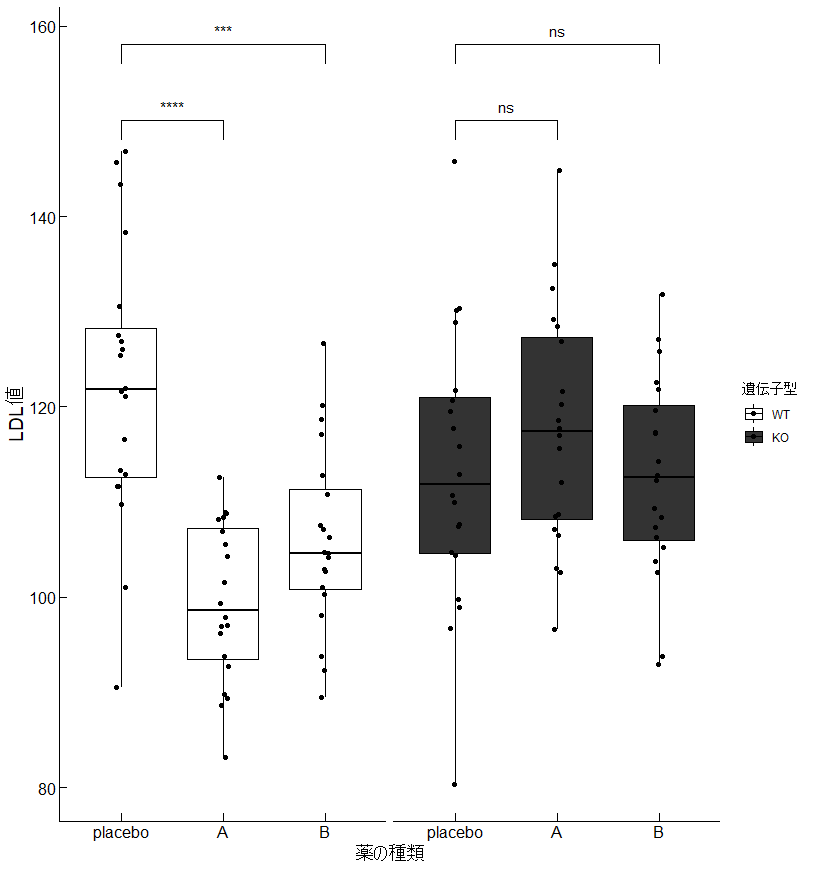

一つずつ解説していきます。
箱ヒゲ図の中心となるコード
ggboxplot(
LDLdata,
x = “drug”, y = “LDL”, color = “group”, fill = “group”,
add = “jitter”, # ドットプロットを追加
facet.by = “group”, # “グループ”で群分け(ファセットの表示)
palette = c(“white”, “gray20”) # 塗りつぶしの色を指定 (WT: 白, KO: 黒に近い灰色)
)
この部分で、箱ヒゲ図をどのデータで描くかについて指示しています。
これが基本の形になります。追加でドットプロットの追加、グループ分け、色の変更を以下でします。
必要がなければこれらはなくてもグラフはかけます。
今回は、コントロール群を白色、介入群を黒に近い灰色にしたため、コントロール群を見やすくするために枠線を黒にしています。
# 枠線を黒に強制
scale_color_manual(values = c(“black”, “black”))
有意差の線を表示する方法
統計的な有意差については、注意を要する形で表示することができます。
stat_compare_means(
comparisons = list(c(“placebo”, “A”), c(“placebo”, “B”)),
method = “t.test”,
label = “p.signif”
)
箱ヒゲ図のレイアウトの調整
labs(x = “薬の種類”, y = “LDL値”, color = “遺伝子型”, fill = “遺伝子型”) +
theme_classic()+
theme(
strip.background = element_blank(), # ファセットタイトルの背景を消す
strip.text = element_blank(), # ファセットタイトルのテキストを消す
axis.line = element_line(color = “black”, size = 0.3, linetype = “solid”),
axis.text = element_text(color = “black”, size = 12),
axis.title = element_text(size = 14),
panel.background = element_blank(), # ファセットの背景を削除
axis.ticks.length = unit(-2, “mm”),
axis.ticks = element_line(color = “black”, size = 0.3),
)
これで見やすい用に調整しています。
axisは軸、lineは線、textは文字、titleはタイトル、などで大きさや色を調整しています。



大変だけど箱ヒゲ図の基本的な描き方がわかったのだ!



ただ注意が必要なのが、P値の有意差を示す線やで。何も考えずにこの描画をすると多重性の問題で本当なら有意差ないのに有意差があるように見えるのがあるでな。
箱ヒゲ図とドットグラフを細かく調整する方法
ここからは、好みに応じて細かい調整をする場合について解説していきます。
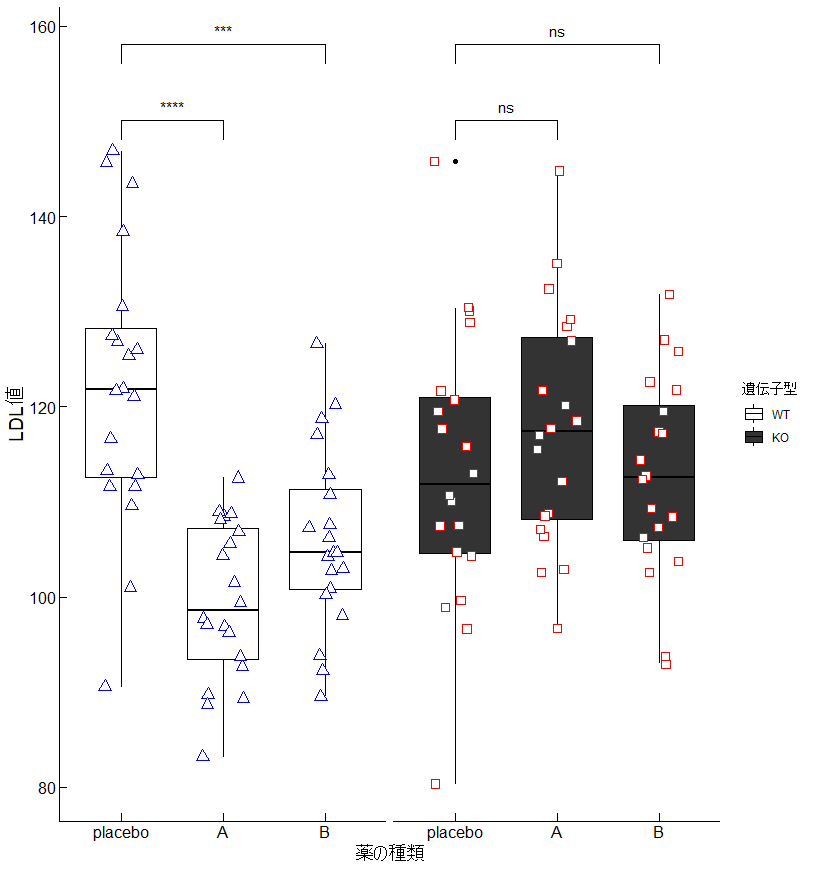
ドットの種類、色、サイズを変える例
ggboxplotの中の『add =”jitter”』では細かいドットの調整ができません!
そのため、この部分を削除して新たにドットを作成する必要があります。
データシートは先程作成したLDLdataファイルを使用します。
library(ggpubr)
# ggboxplotを使用して箱ひげ図を作成
ggboxplot(
LDLdata,
x = "drug", y = "LDL", color = "group", fill = "group",
facet.by = "group", # "グループ"で群分け(ファセットの表示)
palette = c("white", "gray20") # 塗りつぶしの色を指定 (WT: 白, KO: 黒に近い灰色)
) +
# 枠線を黒に強制
scale_color_manual(values = c("black", "black")) +
# 有意差検定の追加
stat_compare_means(
comparisons = list(c("placebo", "A"), c("placebo", "B")),
method = "t.test",
label = "p.signif"
) +
# ラベル設定
labs(x = "薬の種類", y = "LDL値", color = "遺伝子型", fill = "遺伝子型") +
# テーマ設定
theme_classic() +
theme(
strip.background = element_blank(), # ファセットタイトルの背景を消す
strip.text = element_blank(), # ファセットタイトルのテキストを消す
axis.line = element_line(color = "black", size = 0.3, linetype = "solid"),
axis.text = element_text(color = "black", size = 12),
axis.title = element_text(size = 14),
panel.background = element_blank(), # ファセットの背景を削除
axis.ticks.length = unit(-2, "mm"),
axis.ticks = element_line(color = "black", size = 0.3)
) +
# ドットのカスタマイズ (group毎に形状を変更)
geom_jitter(
size = 3, # ドットのサイズを大きく
shape = 24, # ドットの形状を三角形に設定
color = "black", # ドットの枠線を黒に設定
fill = "white", # ドットの塗りつぶしを白に設定
width = 0.2, # 水平のばらつきを少し調整
height = 0.1, # 垂直のばらつきを少し調整
show.legend = FALSE # ドットグラフの凡例の削除
) 前半は『add =”jitter”』を削除した以外変更はありません。
後半にgeom_jitterでドットグラフを棒グラフの上に重ねて表示するようにしています。
ドットのカスタマイズ方法について説明していきます。
一括でサイズなどを変更する場合
geom_jitter(
size = 3, # ドットのサイズを大きく
shape = 24, # ドットの形状を三角形に設定
color = “black”, # ドットの枠線を黒に設定
fill = “white”, # ドットの塗りつぶしを白に設定
width = 0.2, # 水平のばらつきを少し調整
height = 0.1 # 垂直のばらつきを少し調整
show.legend = FALSE # ドットグラフの凡例の削除
)
各種パラメータを好みで変更できます!
ドットの形は以下のように数字が適応されています。
| Shape | 数値 | 形状 |
|---|---|---|
| ● | 19 | 塗りつぶし円 |
| ○ | 1 | 白抜き円 |
| ▲ | 17 | 塗りつぶし三角形 |
| △ | 24 | 白抜き三角形 |
| ■ | 15 | 塗りつぶし正方形 |
| □ | 22 | 白抜き正方形 |
グループ毎に個別にサイズなどを変更したい場合
geom_jitter(
aes(
shape = group, # 形状を group で変更
color = group, # 色を group で変更
size = group # サイズを group で変更
),
fill = "white", # 塗りつぶし色 (形状によって適用される)
width = 0.2, # 水平のばらつきを調整
height = 0.1, # 垂直のばらつきを調整
show.legend = FALSE # ドットグラフの凡例の削除
) +
# 形状のマッピング (WTとKOに対応する形状を指定)
scale_shape_manual(values = c("WT" = 24, "KO" = 22)) + # WT: 三角形, KO: 四角形
# 色のマッピング (WTとKOに対応する色を指定)
scale_color_manual(values = c("WT" = "blue", "KO" = "red")) + # WT: 青, KO: 赤
# サイズのマッピング (WTとKOに対応するサイズを指定)
scale_size_manual(values = c("WT" = 3, "KO" = 4)) # WT: サイズ3, KO: サイズ4aes(・・・)で形状(shape)、色(color)、大きさ(size)をどの群でわけるか指定しています。
今回はgroup列(WT, KO)で分けたいので、『shape = group』などのようにしています。
凡例が箱ヒゲ図と重複して見にくい場合があるので、『show.legend = FALSE』でドットグラフの凡例を削除しています。
これに加えて、scale_(・・・)_manualでそれぞれ形状、色、大きさを個別に指定しています。

棒グラフにドットグラフを組み合わせる方法
先ほどと同じデータシートLDLdataを用いてコードをお示します。
基本的にはggboxplotをggbarplotに変更するだけですが、美しく見えるようにレイアウト調整が必要です。
library(ggpubr)
# ggbarplotを使用して棒グラフを作成
ggbarplot(
LDLdata,
x = "drug", y = "LDL", fill = "group",
facet.by = "group", # ファセット表示
palette = c("white", "gray20"), # 塗りつぶしの色を指定 (WT: 白, KO: 黒に近い灰色)
add = "mean_sd", # 平均と標準偏差を追加
position = position_dodge(width = 0.9) # グループ間の棒を並べる
) +
# 有意差検定の追加
stat_compare_means(
comparisons = list(c("placebo", "A"), c("placebo", "B")),
method = "t.test",
label = "p.signif"
) +
# ラベル設定
labs(x = "薬の種類", y = "LDL値", fill = "遺伝子型") +
# テーマ設定
theme_classic() +
theme(
strip.background = element_blank(), # ファセットタイトルの背景を消す
strip.text = element_blank(), # ファセットタイトルのテキストを消す
axis.line = element_line(color = "black", size = 0.3, linetype = "solid"),
axis.text = element_text(color = "black", size = 12),
axis.title = element_text(size = 14),
panel.background = element_blank(), # ファセットの背景を削除
axis.ticks.length = unit(-2, "mm"),
axis.ticks = element_line(color = "black", size = 0.3),
axis.ticks.x = element_blank(), # x軸のティックを消す
axis.title.x = element_blank(), # x軸のタイトルを消す
axis.line.x = element_blank() # X軸のラインを消す
) +
# ドットのカスタマイズ
geom_jitter(
aes(
shape = group, # 形状を group で変更
color = group # 色を group で変更
),
size = 3, # ドットのサイズ
fill = "white", # 塗りつぶし色
width = 0.2, # 水平のばらつきを調整
height = 0.1, # 垂直のばらつきを調整
show.legend = FALSE # geom_jitter の凡例を非表示
) +
# 形状・色のマッピング
scale_shape_manual(values = c("WT" = 24, "KO" = 22)) +
scale_color_manual(values = c("WT" = "blue", "KO" = "red")) +
# y = 0の実線を引く
geom_hline(yintercept = 0, linetype = "solid", color = "black", size = 0.5)+
# # y軸を0から180までの表示にする
scale_y_continuous(expand = c(0, 0), limits = c(0, 180))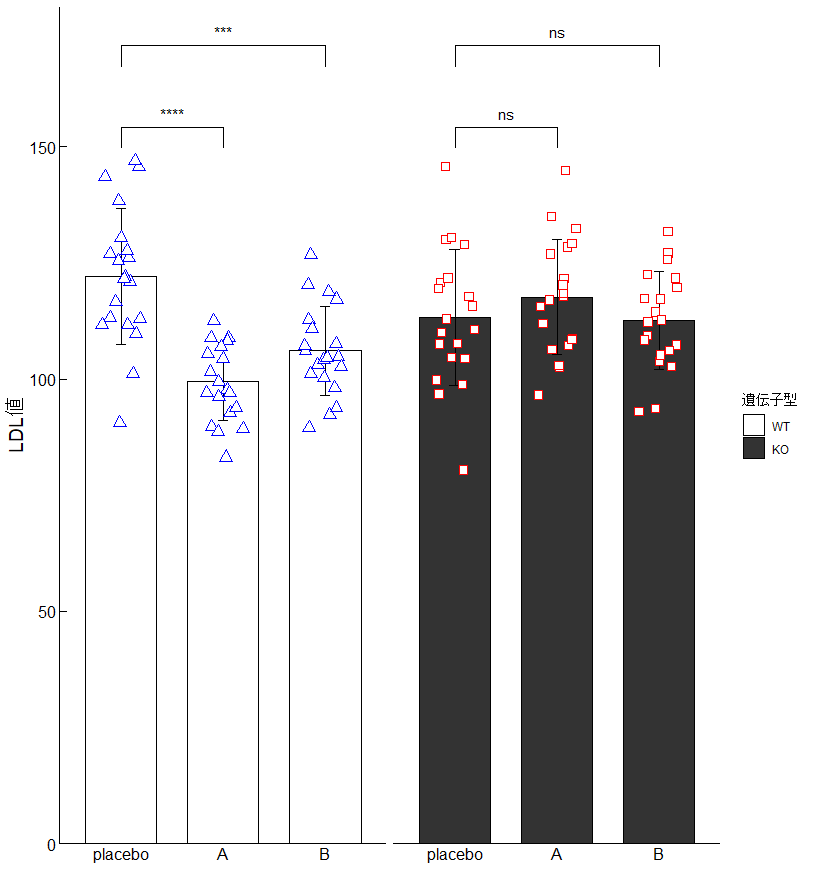

棒グラフでX軸が0のラインになるように調整する方法
theme(
…
…
axis.ticks.x = element_blank(), # x軸のティックを消す
axis.title.x = element_blank(), # x軸のタイトルを消す
axis.line.x = element_blank() # X軸のラインを消す
) +
# y = 0の実線を引く
geom_hline(yintercept = 0, linetype = “solid”, color = “black”, size = 0.5)+
# Y軸のラインを0から表示するようにする
scale_y_continuous(expand = c(0, 0), limits = c(0, 180))
以上のように、まずtheme()の中身で、X軸のティック、タイトル、ラインを消します。
続いて、y = 0の実践を引いて、表示範囲をY軸のライン0からに設定しています。
P値を多重性を補正したadjusted P値でグラフに表示する方法
パッケージ『ggpubr』でのstat_compare_meansでは、多重に統計処理を行った場合のBonferroni法などの補正について対応されておらず、別の手法で解決する方法があります。
ここでは、多重性を調整したP値をグラフに追加する方法について解説します。
データシートについては引き続きLDLdataを使用していきます。
P値を計算したテーブルを作成し、補正したP値をテーブルに組み込む
パッケージ『tidyverse』と『rstatix』をインストールしておいてください。
library(tidyverse)
library(rstatix)
# ttestでのP値の計算
p_values <- LDLdata %>%
group_by(group) %>%
t_test(LDL ~ drug, p.adjust.method = "bonferroni")
# p値のフォーマット関数
format_p_value <- function(p) {
if (p < 0.001) {
return("<0.001")
} else {
return(sprintf("%.3f", p))
}
}
# フォーマットを適用してp値を表示
p_values <- p_values %>%
mutate(adjusted_p_value = sapply(p.adj, format_p_value))LDLdataをグループ毎(WT群、KO群)にわけて、Drug毎のLDLをt_testで計算してます。
このt_testをwilcox_testに変更すれば、Wilcoxonテストを行うことができます。
またbonferroni法以外にも、”holm”, “hochberg”, “hommel”, “bonferroni”, “BH”, “BY”, “fdr”, “none”など他の方法で多重性の調整をすることが可能です。



悩んだらbonferroni(よりP値が出にくい方法)かholm(bonferroniよりP値が出やすい方法)を選んでおけば極論OKなのだ
続いて、P値のフォーマット関数を自作しています。
これは、P<0.001なら『p < 0.001』と表記させ、それ以外の場合は小数点有効数字三桁で表示するようにしています。
最後のスクリプトで、adjusted_p_valueという列をp-valuesというテーブルに追加しています。
最終的には以下のような表になっています。
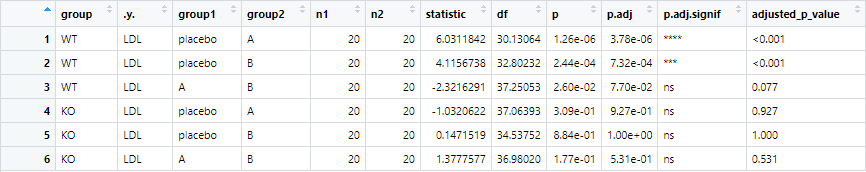

これでajudste_p_valueが作成されました!
補正したP値をグラフに表示する
library(ggpubr)
# ggbarplotを使用して棒グラフを作成
ggbarplot(
LDLdata,
x = "drug", y = "LDL", fill = "group",
facet.by = "group", # ファセット表示
palette = c("white", "gray20"), # 塗りつぶしの色を指定 (WT: 白, KO: 黒に近い灰色)
add = "mean_sd", # 平均と標準偏差を追加
position = position_dodge(width = 0.9) # グループ間の棒を並べる
) +
# 補正したP値をグラフに書き込む
geom_signif(
data = p_values,
aes(xmin = group1, xmax = group2, annotations = adjusted_p_value, y_position = c(150, 160, 170, 150, 160, 170)),
textsize = 6,
tip_length = 0.01,
manual = TRUE
) +
# ラベル設定
labs(x = "薬の種類", y = "LDL値", fill = "遺伝子型") +
# テーマ設定
theme_classic() +
theme(
strip.background = element_blank(), # ファセットタイトルの背景を消す
strip.text = element_blank(), # ファセットタイトルのテキストを消す
axis.line = element_line(color = "black", size = 0.3, linetype = "solid"),
axis.text = element_text(color = "black", size = 12),
axis.title = element_text(size = 14),
panel.background = element_blank(), # ファセットの背景を削除
axis.ticks.length = unit(-2, "mm"),
axis.ticks = element_line(color = "black", size = 0.3),
axis.ticks.x = element_blank(), # x軸のティックを消す
axis.title.x = element_blank(), # x軸のタイトルを消す
axis.line.x = element_blank() # X軸のラインを消す
) +
# ドットのカスタマイズ
geom_jitter(
aes(
shape = group, # 形状を group で変更
color = group # 色を group で変更
),
size = 3, # ドットのサイズ
fill = "white", # 塗りつぶし色
width = 0.2, # 水平のばらつきを調整
height = 0.1, # 垂直のばらつきを調整
show.legend = FALSE # geom_jitter の凡例を非表示
) +
# 形状・色のマッピング
scale_shape_manual(values = c("WT" = 24, "KO" = 22)) +
scale_color_manual(values = c("WT" = "blue", "KO" = "red")) +
# y = 0の実線を引く
geom_hline(yintercept = 0, linetype = "solid", color = "black", size = 0.5)+
# y軸を0から180までの表示にする
scale_y_continuous(expand = c(0, 0), limits = c(0, 180))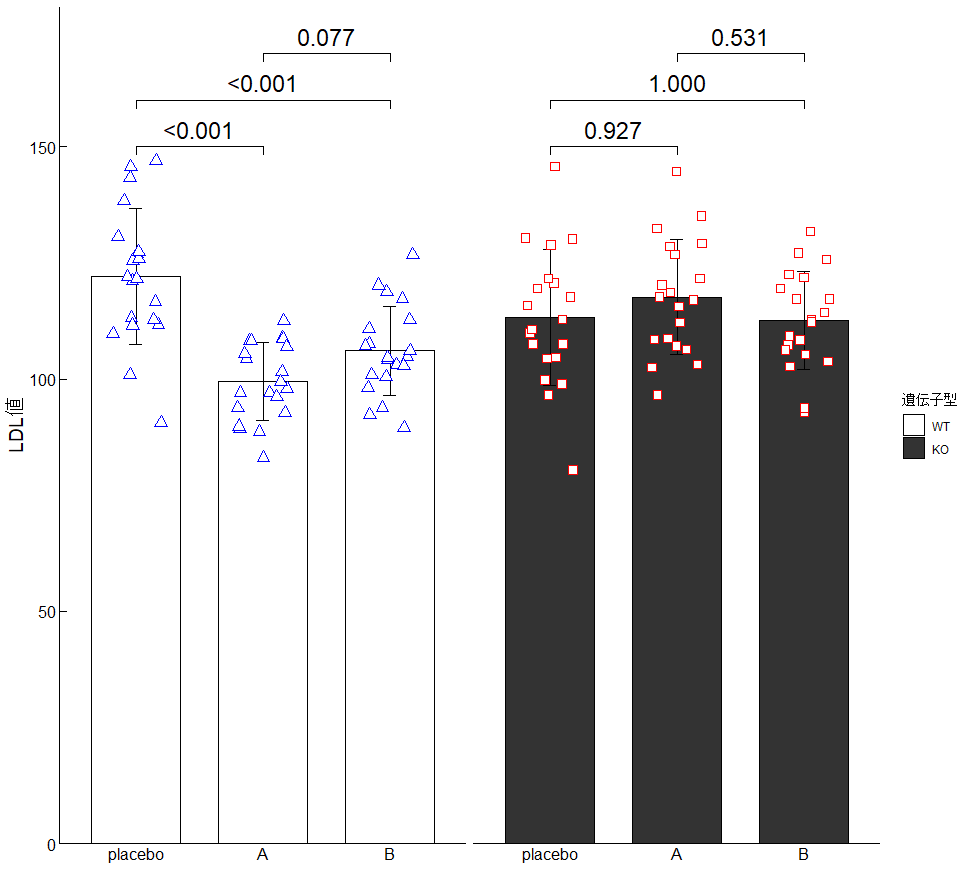

今までのstat_compare_meansの代わりに以下のコードを入力しています。
# 補正したP値をグラフに書き込む geom_signif( data = p_values, aes(xmin = group1, xmax = group2, annotations = adjusted_p_value, y_position = c(150, 160, 170, 150, 160, 170)), textsize = 6, tip_length = 0.01, manual = TRUE ) +
geom_signif()で有意差のグラフをかけるように指示しています。
その有意差のデータをどのデータシートから持ってくるのかdata = データシート名で指示しています。
自動で認識しないので以下のコードで細かく指示しています。
aes(xmin = 有意差ラインの左端になる列名, xmax = 有意差ラインの右端になる列名, annotations = 有意差として表示したいP値, y_position = c(数値1, 数値2, 数値3, …)
先ほどのp_valuesの図を見ながら説明します。


有意差ラインの左端はgroup1, 右端はgroup2, P値はadjusted_p_valueでグラフを書いています。
y_positionについては自動で高さを決定することができません。
そのため、グラフのY軸に応じて手動で入力して微調整する必要があります。
textsize = 6,
tip_length = 0.01,
manual = TRUE
これで、P値の文字のサイズ、有意差ラインの端の縦線の長さを指示しています。
manual = TRUEはこれまでの細かい指示を有効にするためのコードです。
これで補正したP値をグラフに表示することができました!!
まとめ
ドットプロットと箱ヒゲ図を組み合わせたグラフの描き方をメインに解説しました!
少しでもグラフ作成のお役に立てれば幸いです!!










コメント