こんにちは。ほしのはやしです。
臨床系のデータを扱う場合、カプランマイヤー曲線が必須になること、多いですよね。
このページではR studioでカプランマイヤー曲線(生存曲線など)を書く方法と統計解析について解説します!
カプランマイヤー曲線(生存曲線など)
カプランマイヤー曲線を使う場合
ある集団において、イベント(死亡、退院、故障など)までの時間を可視化したい場合です。
例えば、大腸癌の化学療法でProgression free survival(PFS)をみたときは、イベントを病勢の進行と定義しています。
他には、ICU患者である治療をしてICU在室日数をみたときは、イベントをICU退室と定義しています。
 星柴くん
星柴くん全生存期間をみたい場合は、死亡がイベントになっていることが多いんだね!
このように、時間経過でイベントの発生がどうなっているかを一目で確認するのに便利な図になります。
打ち切り(censoring)ってどういう意味?
目的のイベント以外の理由で時間を追跡できなくなった場合に打ち切り(censoring)といいます。
例えば、大腸癌の化学療法でPFSをみたくて、イベントを病勢の進行とした場合に、途中で転院したとか、他の病気でなくなったとか、病勢の進行のタイミングがわからなくなった時間を打ち切り日として扱います。
この打ち切りについて、一つ注意が必要なのが「競合リスク」の存在です。
臨床研究では、競合リスクは非常に重要な概念です。
例えば、ある抗がん剤の治療効果を評価する場合、がんによる死亡だけでなく、治療に伴う副作用による死亡も起こり得ます。この時、がんによる死亡が起きる前に、副作用によって患者さんが亡くなってしまうと、その患者さんについては抗がん剤の治療効果を評価することができなくなります。つまり、副作用による死亡が「競合リスク」となるのです。このような競合リスクの存在を考慮せずに分析を行うと、治療効果を過小評価したり、逆に過大評価したりする可能性があります。
競合リスク分析についての詳しい手法については以下のページを参考にしてください!


右側打ち切り、左側打ち切りって?
ずばり言いましょう。
臨床研究をする場合は、ほとんど右側打ち切りだけなので気にしなくて大丈夫です!
右側打ち切りとは、観察開始より後で打ち切りが起きた場合で、先程の例や観察期間終了が含まれます。
左側打ち切りとは、観察開始より前で打ち切りが起きた場合で、例えばある薬を投与してからの心筋梗塞発生までの日数を調べたい場合、ランダム化して薬を投与するまでに心筋梗塞発症した場合になります。
R studioにおける打ち切りデータの入力の仕方
統計ソフトによって打ち切りデータの入力が異なりますので注意が必要です。
R studioの場合は、0(打ち切り(右側))、1(イベント発生)、3(左側打ち切り)、となっていますので、データ入力の場合は、0か1を入力しておくと良いです。
具体的なコード(R studio)
とても便利なsurvminerというパッケージをインストールしておいてください。
パッケージのインストール方法はこちら!


テーブル名:datasheet1
| Age | Sex | PFS_time | PFS_censore | Stage | Intervention |
|---|---|---|---|---|---|
| 65 | M | 78 | 1 | I | Placebo |
| 70 | F | 80 | 1 | IV | DrugA |
| 62 | M | 145 | 0 | II | DrugA |
| 81 | M | 50 | 1 | III | DrugA |
| 76 | F | 55 | 1 | I | Placebo |
| 77 | F | 123 | 1 | IV | Placebo |
| … | … | … | … | … | … |
library(survival)
library(survminer)
fit1 <- survfit(Surv(PFS_time, PFS_censore) ~ Intervention, datasheet1)
ggsurvplot(fit1)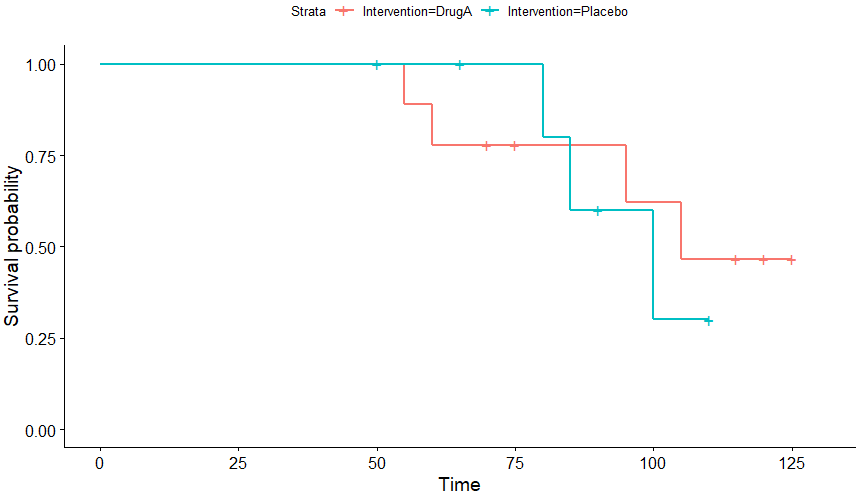

これがデフォルトになります。
より論文に適した形にしたい場合は、以下のように微調整します。
library(survival)
library(survminer)
fit1 <- survfit(Surv(PFS_time, PFS_censore) ~ Intervention, datasheet1)
ggsurvplot(
fit1,
pval = TRUE,
risk.table = TRUE,
palette = c("blue","red"),
censor.shape = "|",
legend.title = "Intervention",
legend.lab = c("Drug A", "Placebo"),
xlab = "Days since intervention",
ylab = "Probability of PFS"
)




ここで出るP値はログランク検定で計算されたものになるよ
Medianと95%CIは、先程のコードのあとにfit1とうつだけで確認できます!
fit1

イベントまでの期間の統計解析(生存期間の統計解析)
統計で使う検定は?
ログランク検定(Log-rank検定)になります。
これは2群以上の比較において使えるノンパラメトリック検定になります。
具体的なコード(R studio)
library(survival)
library(survminer)
res2 <- survdiff(Surv(PFS_time, PFS_censore) ~ Intervention, datasheet1)
print(res2, digit = 4)

最後のp値が求められた値になります。
print(結果のデータ, digit = 数字)で小数点何桁まで表示するか決定できます!
3群以上を比較する場合(pairwise_survdiff)
3群以上の場合は、False discovery rate(FDR)の調整が必要になります。
以下のコードを覚えて守られば大丈夫です!
先程と同じ表を使います。
library(survival)
library(survminer)
res3 <- pairwise_survdiff(Surv(PFS_time, PFS_censore) ~ Intervention, datasheet1, p.adjust.method = "holm")
print(res3, digit = 4)カプランマイヤー曲線でサブグループ解析
カテゴリー因子でサブグループ解析
すでにカテゴリー化されているSexについてサブグループ解析をする方法です。
テーブル名:datasheet1
| Age | Sex | PFS_time | PFS_censore | Stage | Intervention |
|---|---|---|---|---|---|
| 65 | M | 78 | 1 | I | Placebo |
| 70 | F | 80 | 1 | IV | DrugA |
| 62 | M | 145 | 0 | II | DrugA |
| 81 | M | 50 | 1 | III | DrugA |
| 76 | F | 55 | 1 | I | Placebo |
| 77 | F | 123 | 1 | IV | Placebo |
| … | … | … | … | … | … |
library(survival)
library(survminer)
fit1 <- survfit(Surv(PFS_time, PFS_censore) ~ Intervention, datasheet1)
ggsurvplot(
fit1,
palette = c("blue", "red"),
censor.shape = "|",
legend.title = "Intervention",
legend.lab = c("Drug A", "Placebo"),
xlab = "Days since intervention",
ylab = "Probability of PFS",
facet.by = "Sex" # ここにサブグループになる変数を指定
)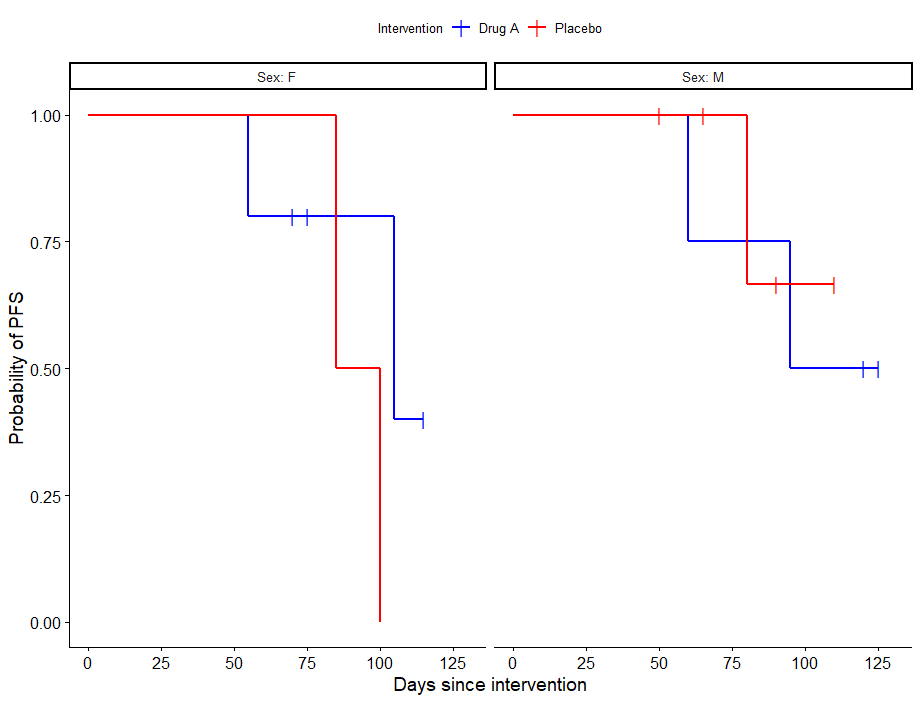

survminerのパッケージを使って、最後にfacet.by =”サブグループ解析したい列名”を入れることで簡単にできます!
連続変数(年齢など)を層別化してサブグループ解析
年齢など連続した数値をサブグループ解析するためには、適当なグループに分類する必要があります。
年齢を例に出してコードを記載します。
datasheet1$Age_group <- ifelse(datasheet1$Age >= 65, "≥ 65 yr", "< 65 yr")新しく、年齢を65歳以上と65歳未満で分けてAge_groupというのをdatasheet1に追加するスクリプトです。
これを元に層別化してカプランマイヤーを書きます。
library(survival)
library(survminer)
fit1 <- survfit(Surv(PFS_time, PFS_censore) ~ Intervention, datasheet1)
ggsurvplot(
fit1,
palette = c("blue", "red"),
censor.shape = "|",
legend.title = "Intervention",
legend.lab = c("Drug A", "Placebo"),
xlab = "Days since intervention",
ylab = "Probability of PFS",
facet.by = "Age_group" # ここにサブグループになる変数を指定
)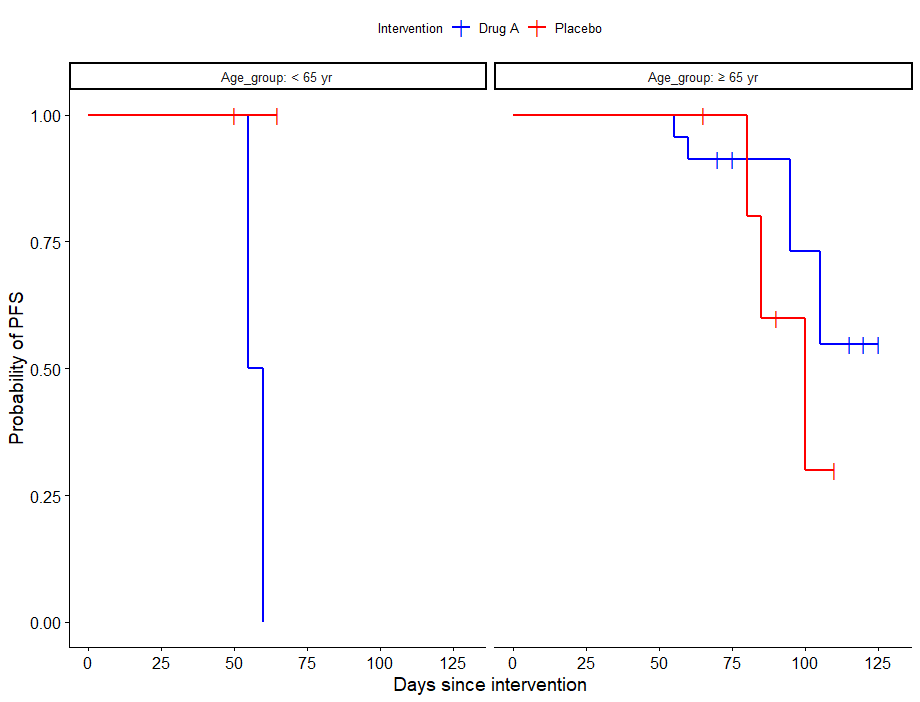




これで層別化したカプランマイヤー曲線をかけるね!
まとめ
カプランマイヤー曲線の書き方とログランク検定について解説しました。
少しでもお役に立てれば幸いです。










コメント